课程名称:菜菜的sklearn机器学习
课程地址:跳转
sklearn中的数据预处理和特征工程
2 数据预处理 Preprocessing & Impute
2.1 数据无量纲化
在机器学习算法实践中,我们往往有着将不同规格的数据转换到同一规格,或不同分布的数据转换到某个特定分布
的需求,这种需求统称为将数据“无量纲化”。譬如梯度和矩阵为核心的算法中,譬如逻辑回归,支持向量机,神经
网络,无量纲化可以加快求解速度;而在距离类模型,譬如K近邻,K-Means聚类中,无量纲化可以帮我们提升模
型精度,避免某一个取值范围特别大的特征对距离计算造成影响。(一个特例是决策树和树的集成算法们,对决策
树我们不需要无量纲化,决策树可以把任意数据都处理得很好。)
数据的无量纲化可以是线性的,也可以是非线性的。线性的无量纲化包括中心化(Zero-centered或者Meansubtraction)处理和缩放处理(Scale)。中心化的本质是让所有记录减去一个固定值,即让数据样本数据平移到某个位置。缩放的本质是通过除以一个固定值,将数据固定在某个范围之中,取对数也算是一种缩放处理
- preprocessing.MinMaxScaler
当数据(x)按照最小值中心化后,再按极差(最大值 - 最小值)缩放,数据移动了最小值个单位,并且会被收敛到
[0,1]之间,而这个过程,就叫做数据归一化(Normalization,又称Min-Max Scaling)。注意,Normalization是归
一化,不是正则化,真正的正则化是regularization,不是数据预处理的一种手段。归一化之后的数据服从正态分
布,公式如下:
$$x^{*}=\frac{x-\min (x)}{\max (x)-\min (x)}$$
在sklearn当中,我们使用preprocessing.MinMaxScaler来实现这个功能。MinMaxScaler有一个重要参数,
feature_range,控制我们希望把数据压缩到的范围,默认是[0,1]。
from sklearn.preprocessing import MinMaxScaler
data = [[-1, 2], [-0.5, 6], [0, 10], [1, 18]]
#不太熟悉numpy的小伙伴,能够判断data的结构吗?
#如果换成表是什么样子?
import pandas as pd
pd.DataFrame(data)
#实现归一化
scaler = MinMaxScaler() #实例化
scaler = scaler.fit(data) #fit,在这里本质是生成min(x)和max(x)
result = scaler.transform(data) #通过接口导出结果
result
result_ = scaler.fit_transform(data) #训练和导出结果一步达成
scaler.inverse_transform(result) #将归一化后的结果逆转
#使用MinMaxScaler的参数feature_range实现将数据归一化到[0,1]以外的范围中
data = [[-1, 2], [-0.5, 6], [0, 10], [1, 18]]
scaler = MinMaxScaler(feature_range=[5,10]) #依然实例化
result = scaler.fit_transform(data) #fit_transform一步导出结果
result
#当X中的特征数量非常多的时候,fit会报错并表示,数据量太大了我计算不了
#此时使用partial_fit作为训练接口
#scaler = scaler.partial_fit(data)BONUS:用numpy实现
import numpy as np
X = np.array([[-1, 2], [-0.5, 6], [0, 10], [1, 18]])
#归一化
X_nor = (X - X.min(axis=0)) / (X.max(axis=0) - X.min(axis=0))
X_nor
#逆转归一化
X_returned = X_nor * (X.max(axis=0) - X.min(axis=0)) + X.min(axis=0)
X_returned- preprocessing.StandardScaler
当数据(x)按均值(μ)中心化后,再按标准差(σ)缩放,数据就会服从为均值为0,方差为1的正态分布(即标准正态分
布),而这个过程,就叫做数据标准化(Standardization,又称Z-score normalization),公式如下:
$$x^{*}=\frac{x-\mu}{\sigma}$$
from sklearn.preprocessing import StandardScaler
data = [[-1, 2], [-0.5, 6], [0, 10], [1, 18]]
scaler = StandardScaler() #实例化
scaler.fit(data) #fit,本质是生成均值和方差
scaler.mean_ #查看均值的属性mean_
scaler.var_ #查看方差的属性var_
x_std = scaler.transform(data) #通过接口导出结果
x_std.mean() #导出的结果是一个数组,用mean()查看均值
x_std.std() #用std()查看方差
scaler.fit_transform(data) #使用fit_transform(data)一步达成结果
scaler.inverse_transform(x_std) #使用inverse_transform逆转标准化对于StandardScaler和MinMaxScaler来说,空值NaN会被当做是缺失值,在fit的时候忽略,在transform的时候
保持缺失NaN的状态显示。并且,尽管去量纲化过程不是具体的算法,但在fit接口中,依然只允许导入至少二维数
组,一维数组导入会报错。通常来说,我们输入的X会是我们的特征矩阵,现实案例中特征矩阵不太可能是一维所
以不会存在这个问题
- StandardScaler和MinMaxScaler选哪个?
看情况。大多数机器学习算法中,会选择StandardScaler来进行特征缩放,因为MinMaxScaler对异常值非常敏
感。在PCA,聚类,逻辑回归,支持向量机,神经网络这些算法中,StandardScaler往往是最好的选择。
MinMaxScaler在不涉及距离度量、梯度、协方差计算以及数据需要被压缩到特定区间时使用广泛,比如数字图像
处理中量化像素强度时,都会使用MinMaxScaler将数据压缩于[0,1]区间之中。
建议先试试看StandardScaler,效果不好换MinMaxScaler。
除了StandardScaler和MinMaxScaler之外,sklearn中也提供了各种其他缩放处理(中心化只需要一个pandas广
播一下减去某个数就好了,因此sklearn不提供任何中心化功能)。比如,在希望压缩数据,却不影响数据的稀疏
性时(不影响矩阵中取值为0的个数时),我们会使用MaxAbsScaler;在异常值多,噪声非常大时,我们可能会选
用分位数来无量纲化,此时使用RobustScaler。更多详情请参考以下列表

2.2 缺失值
机器学习和数据挖掘中所使用的数据,永远不可能是完美的。很多特征,对于分析和建模来说意义非凡,但对于实
际收集数据的人却不是如此,因此数据挖掘之中,常常会有重要的字段缺失值很多,但又不能舍弃字段的情况。因
此,数据预处理中非常重要的一项就是处理缺失值。 在这里,我们使用从泰坦尼克号提取出来的数据,这个数据有三个特征,一个数值型,两个字符型,标签也是字符型。从这里开始,我们就使用这个数据给大家作为例子,让大家慢慢熟悉sklearn中数据预处理的各种方式
。
import os
import pandas as pd
data = pd.read_csv('./data/Narrativedata.csv',index_col=0) # index_col=0的意思是:将第0列作为索引
data.head()#默认显示前5行数据| Age | Sex | Embarked | Survived | |
|---|---|---|---|---|
| 0 | 22.0 | male | S | No |
| 1 | 38.0 | female | C | Yes |
| 2 | 26.0 | female | S | Yes |
| 3 | 35.0 | female | S | Yes |
| 4 | 35.0 | male | S | No |
data.info()<class 'pandas.core.frame.DataFrame'>
Int64Index: 891 entries, 0 to 890
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Age 714 non-null float64
1 Sex 891 non-null object
2 Embarked 889 non-null object
3 Survived 891 non-null object
dtypes: float64(1), object(3)
memory usage: 67.1+ KB- impute.SimpleImputer
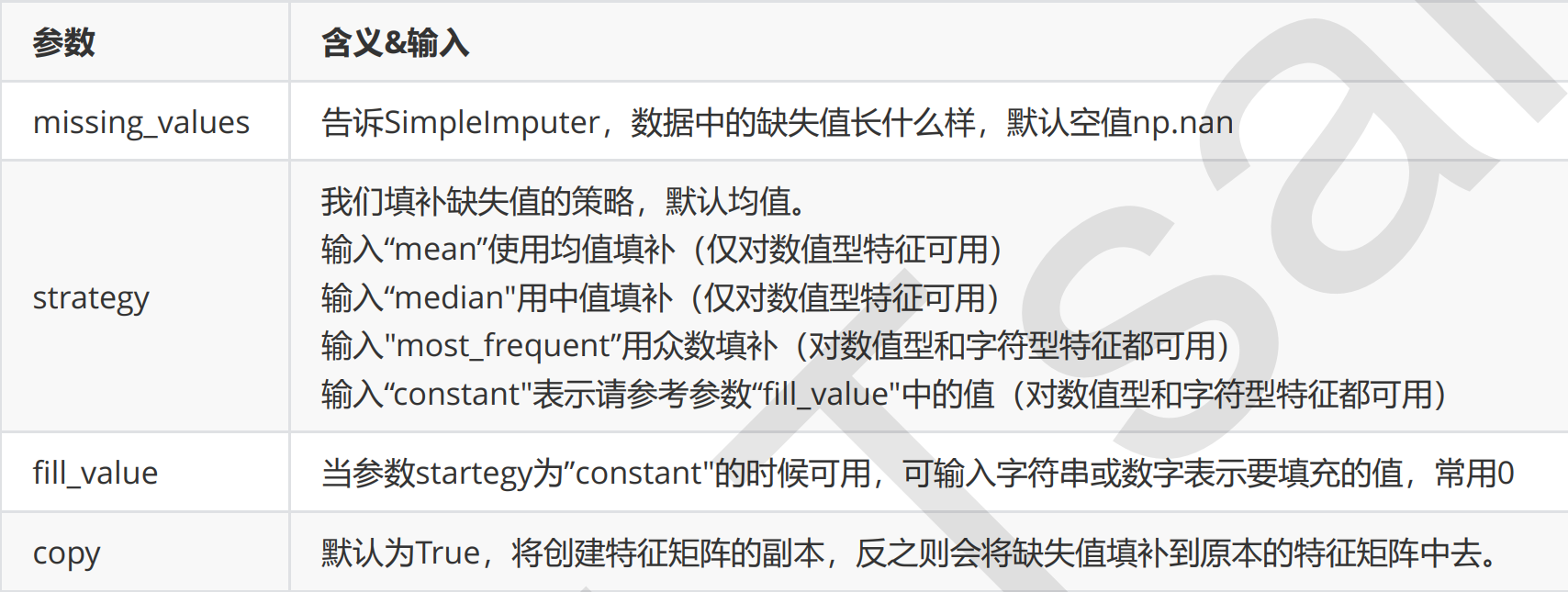
class sklearn.impute.SimpleImputer (missing_values=nan, strategy=’mean’, fill_value=None, verbose=0,copy=True)
在讲解随机森林的案例时,我们用这个类和随机森林回归填补了缺失值,对比了不同的缺失值填补方式对数据的影
响。这个类是专门用来填补缺失值的。它包括四个重要参数:
data.info()
#填补年龄
Age = data.loc[:,"Age"].values.reshape(-1,1) #sklearn当中特征矩阵必须是二维
Age[:20]
from sklearn.impute import SimpleImputer
imp_mean = SimpleImputer() #实例化,默认均值填补
imp_median = SimpleImputer(strategy="median") #用中位数填补
imp_0 = SimpleImputer(strategy="constant",fill_value=0) #用0填补
imp_mean = imp_mean.fit_transform(Age) #fit_transform一步完成调取结果
imp_median = imp_median.fit_transform(Age)
imp_0 = imp_0.fit_transform(Age)
imp_mean[:20]
imp_median[:20]
imp_0[:20]
#在这里我们使用中位数填补Age
data.loc[:,"Age"] = imp_median
data.info()
#使用众数填补Embarked
Embarked = data.loc[:,"Embarked"].values.reshape(-1,1)
imp_mode = SimpleImputer(strategy = "most_frequent")
data.loc[:,"Embarked"] = imp_mode.fit_transform(Embarked)
data.info()<class 'pandas.core.frame.DataFrame'>
Int64Index: 891 entries, 0 to 890
Data columns (total 4 columns):
# Column Non-Null Count Dtype
--- ------ -------------- -----
0 Age 891 non-null float64
1 Sex 891 non-null object
2 Embarked 891 non-null object
3 Survived 891 non-null object
dtypes: float64(1), object(3)
memory usage: 67.1+ KBBONUS:用Pandas和Numpy进行填补其实更加简单
import pandas as pd
data = pd.read_csv(r"C:\work\learnbetter\micro-class\week 3 Preprocessing\Narrativedata.csv",index_col=0)
data.head()
data.loc[:,"Age"] = data.loc[:,"Age"].fillna(data.loc[:,"Age"].median())
#.fillna 在DataFrame里面直接进行填补
data.dropna(axis=0,inplace=True)#inplace开启原地操作
#.dropna(axis=0)删除所有有缺失值的行,.dropna(axis=1)删除所有有缺失值的列
#参数inplace,为True表示在原数据集上进行修改,为False表示生成一个复制对象,不修改原数据,默认False
# _data_ = data_.drop(axis=0,inplace=False)2.3 处理分类型特征:编码与哑变量
在机器学习中,大多数算法,譬如逻辑回归,支持向量机SVM,k近邻算法等都只能够处理数值型数据,不能处理
文字,在sklearn当中,除了专用来处理文字的算法,其他算法在fit的时候全部要求输入数组或矩阵,也不能够导
入文字型数据(其实手写决策树和普斯贝叶斯可以处理文字,但是sklearn中规定必须导入数值型)。
然而在现实中,许多标签和特征在数据收集完毕的时候,都不是以数字来表现的。比如说,学历的取值可以是[“小
学”,“初中”,“高中”,”大学”],付费方式可能包含[“支付宝”,“现金”,“微信”]等等。在这种情况下,为了让数据适
应算法和库,我们必须将数据进行编码,即是说,将文字型数据转换为数值型
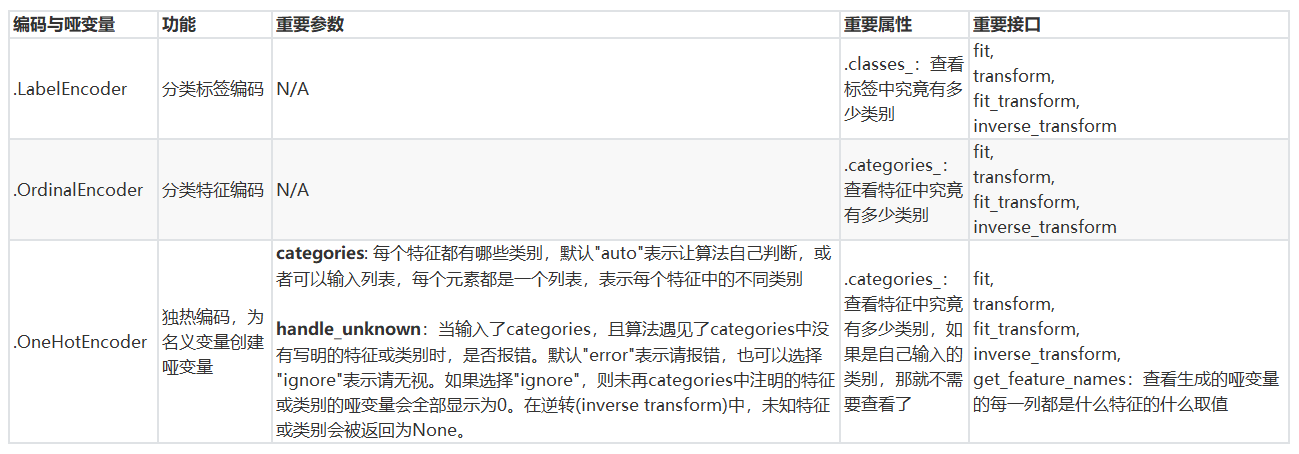
- preprocessing.LabelEncoder:标签专用,能够将分类转换为分类数值
from sklearn.preprocessing import LabelEncoder
y = data_.iloc[:,-1] #要输入的是标签,不是特征矩阵,所以允许一维
le = LabelEncoder() #实例化
le = le.fit(y) #导入数据
label = le.transform(y) #transform接口调取结果
le.classes_ #属性.classes_查看标签中究竟有多少类别
label #查看获取的结果label
le.fit_transform(y) #也可以直接fit_transform一步到位
le.inverse_transform(label) #使用inverse_transform可以逆转
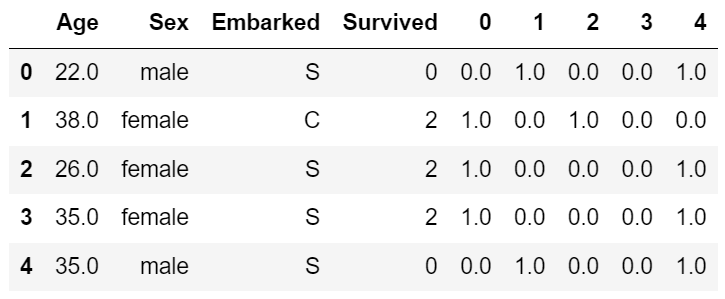
#如果不需要教学展示的话我会这么写:
from sklearn.preprocessing import LabelEncoder
data.iloc[:,-1] = LabelEncoder().fit_transform(data.iloc[:,-1])| Age | Sex | Embarked | Survived | |
|---|---|---|---|---|
| 0 | 22.0 | male | S | 0 |
| 1 | 38.0 | female | C | 2 |
| 2 | 26.0 | female | S | 2 |
| 3 | 35.0 | female | S | 2 |
| 4 | 35.0 | male | S | 0 |
- preprocessing.OrdinalEncoder:特征专用,能够将分类特征转换为分类数值
from sklearn.preprocessing import OrdinalEncoder
#接口categories_对应LabelEncoder的接口classes_,一模一样的功能
data_ = data.copy()
data_.head()
OrdinalEncoder().fit(data_.iloc[:,1:-1]).categories_
data_.iloc[:,1:-1] = OrdinalEncoder().fit_transform(data_.iloc[:,1:-1])
data_.head()| Age | Sex | Embarked | Survived | |
|---|---|---|---|---|
| 0 | 22.0 | 1.0 | 2.0 | 0 |
| 1 | 38.0 | 0.0 | 0.0 | 2 |
| 2 | 26.0 | 0.0 | 2.0 | 2 |
| 3 | 35.0 | 0.0 | 2.0 | 2 |
| 4 | 35.0 | 1.0 | 2.0 | 0 |
- preprocessing.OneHotEncoder:独热编码,创建哑变量
我们刚才已经用OrdinalEncoder把分类变量Sex和Embarked都转换成数字对应的类别了。在舱门Embarked这一
列中,我们使用[0,1,2]代表了三个不同的舱门,然而这种转换是正确的吗?
我们来思考三种不同性质的分类数据:
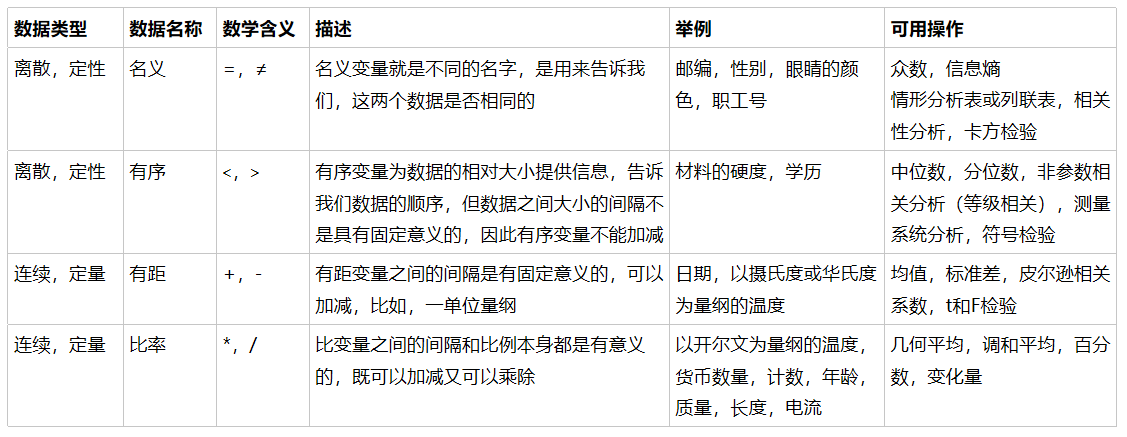
1) 舱门(S,C,Q)
三种取值S,C,Q是相互独立的,彼此之间完全没有联系,表达的是S≠C≠Q的概念。这是名义变量。
2) 学历(小学,初中,高中)
三种取值不是完全独立的,我们可以明显看出,在性质上可以有高中>初中>小学这样的联系,学历有高低,但是学
历取值之间却不是可以计算的,我们不能说小学 + 某个取值 = 初中。这是有序变量。
3) 体重(>45kg,>90kg,>135kg)
各个取值之间有联系,且是可以互相计算的,比如120kg - 45kg = 90kg,分类之间可以通过数学计算互相转换。这
是有距变量。
然而在对特征进行编码的时候,这三种分类数据都会被我们转换为[0,1,2],这三个数字在算法看来,是连续且可以
计算的,这三个数字相互不等,有大小,并且有着可以相加相乘的联系。所以算法会把舱门,学历这样的分类特
征,都误会成是体重这样的分类特征。这是说,我们把分类转换成数字的时候,忽略了数字中自带的数学性质,所
以给算法传达了一些不准确的信息,而这会影响我们的建模。
类别OrdinalEncoder可以用来处理有序变量,但对于名义变量,我们只有使用哑变量的方式来处理,才能够尽量
向算法传达最准确的信息:

这样的变化,让算法能够彻底领悟,原来三个取值是没有可计算性质的,是“有你就没有我”的不等概念。在我们的
数据中,性别和舱门,都是这样的名义变量。因此我们需要使用独热编码,将两个特征都转换为哑变量。
data.head()
from sklearn.preprocessing import OneHotEncoder
X = data.iloc[:,1:-1]
enc = OneHotEncoder(categories='auto').fit(X)
result = enc.transform(X).toarray()
result
#依然可以直接一步到位,但为了给大家展示模型属性,所以还是写成了三步
OneHotEncoder(categories='auto').fit_transform(X).toarray()
#依然可以还原
pd.DataFrame(enc.inverse_transform(result))
enc.get_feature_names_out()#返回每一个经过哑变量后生成稀疏矩阵列的名字
result
result.shape
#axis=1,表示跨行进行合并,也就是将两表左右相连,如果是axis=0,就是将量表上下相连
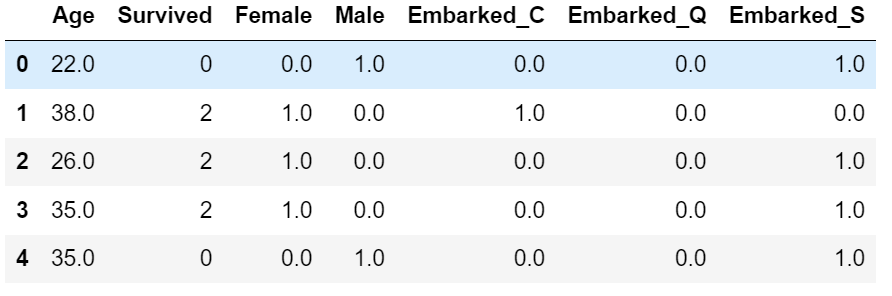
newdata = pd.concat([data,pd.DataFrame(result)],axis=1)
newdata.head()
newdata.drop(["Sex","Embarked"],axis=1,inplace=True)#原地操作,删除这两列
newdata.columns = ["Age","Survived","Female","Male","Embarked_C","Embarked_Q","Embarked_S"]
newdata.head()将特征one-hot表与原来的表进行拼接(concat)
最后再删除原表中的特征列,并且重新修改特征列名称
特征可以做哑变量,标签也可以吗?可以,使用类sklearn.preprocessing.LabelBinarizer可以对做哑变量,许多算
法都可以处理多标签问题(比如说决策树),但是这样的做法在现实中不常见,因此我们在这里就不赘述了
BONUS:数据类型以及常用的统计量
2.4 处理连续型特征:二值化与分段
- sklearn.preprocessing.Binarizer
根据阈值将数据二值化(将特征值设置为0或1),用于处理连续型变量。大于阈值的值映射为1,而小于或等于阈
值的值映射为0。默认阈值为0时,特征中所有的正值都映射到1。二值化是对文本计数数据的常见操作,分析人员
可以决定仅考虑某种现象的存在与否。它还可以用作考虑布尔随机变量的估计器的预处理步骤(例如,使用贝叶斯
设置中的伯努利分布建模)。
#将年龄二值化
data_2 = data.copy()
from sklearn.preprocessing import Binarizer
X = data_2.iloc[:,0].values.reshape(-1,1) #类为特征专用,所以不能使用一维数组
transformer = Binarizer(threshold=30).fit_transform(X)
transformer- preprocessing.KBinsDiscretizer
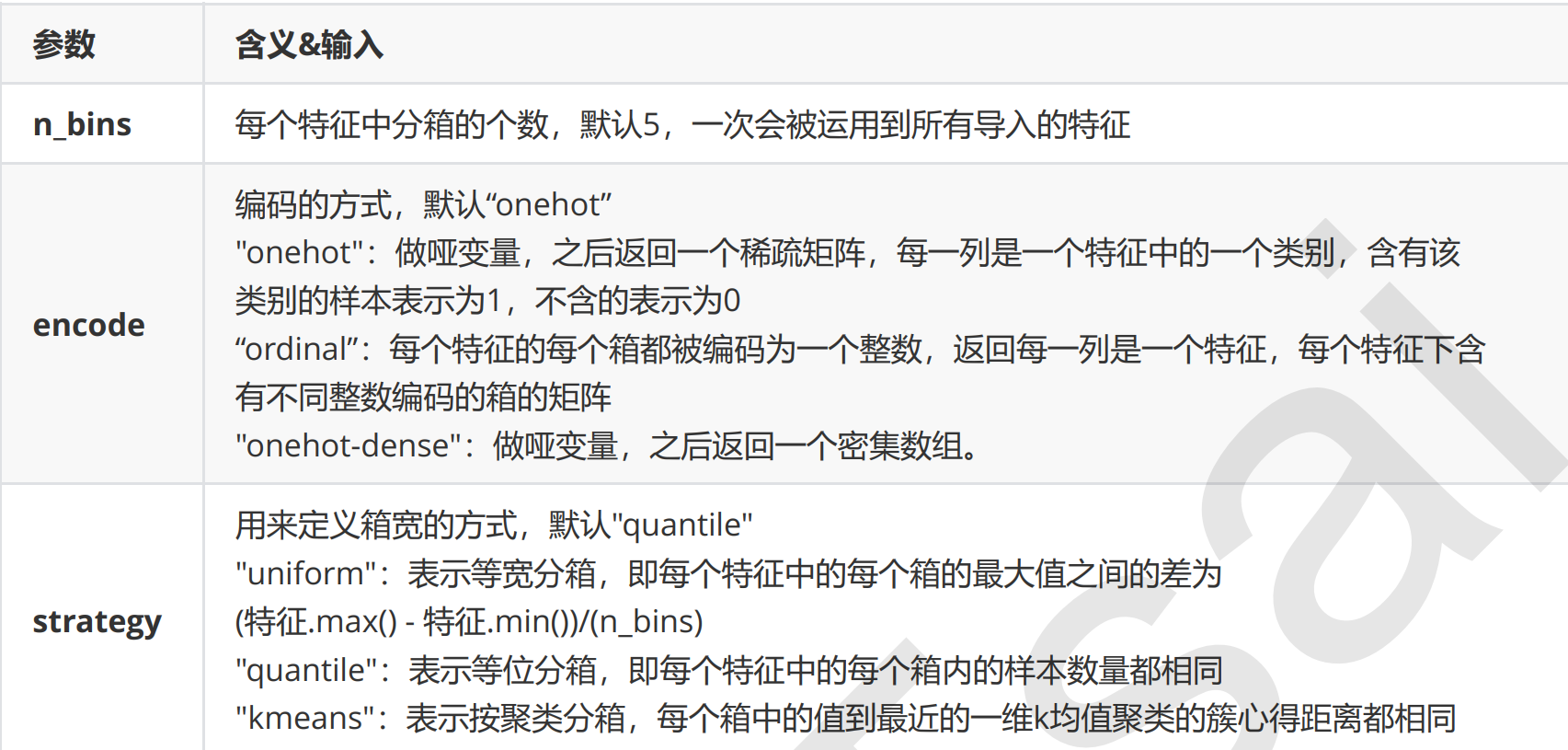
这是将连续型变量划分为分类变量的类,能够将连续型变量排序后按顺序分箱后编码。总共包含三个重要参数:
from sklearn.preprocessing import KBinsDiscretizer
X = data.iloc[:,0].values.reshape(-1,1)
est = KBinsDiscretizer(n_bins=3, encode='ordinal', strategy='uniform')
est.fit_transform(X)
#查看转换后分的箱:变成了一列中的三箱
set(est.fit_transform(X).ravel())
est = KBinsDiscretizer(n_bins=3, encode='onehot', strategy='uniform')
#查看转换后分的箱:变成了哑变量
est.fit_transform(X).toarray()3 特征选择 feature_selection
当数据预处理完成后,我们就要开始进行特征工程了。

在做特征选择之前,有三件非常重要的事:**跟数据提供者开会!跟数据提供者开会!跟数据提供者开会!
**一定要抓住给你提供数据的人,尤其是理解业务和数据含义的人,跟他们聊一段时间。技术能够让模型起飞,前提
是你和业务人员一样理解数据。所以特征选择的第一步,其实是根据我们的目标,用业务常识来选择特征。来看完
整版泰坦尼克号数据中的这些特征:
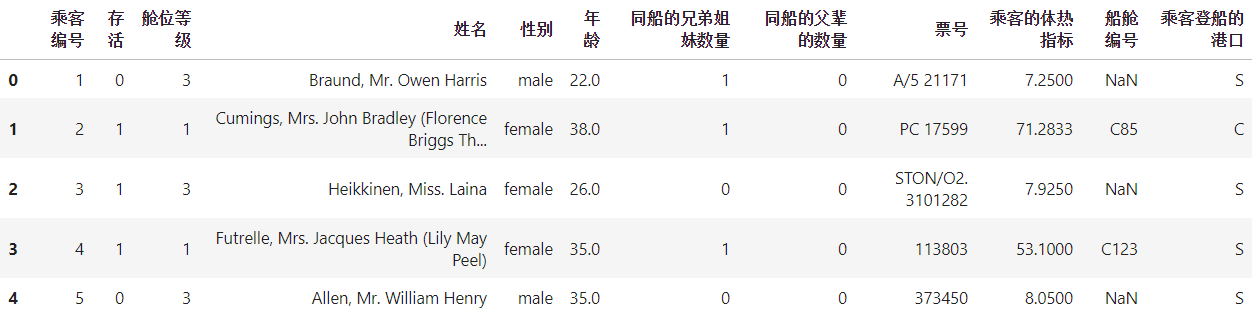
其中是否存活是我们的标签。很明显,以判断“是否存活”为目的,票号,登船的舱门,乘客编号明显是无关特征,
可以直接删除。姓名,舱位等级,船舱编号,也基本可以判断是相关性比较低的特征。性别,年龄,船上的亲人数
量,这些应该是相关性比较高的特征。
所以,特征工程的第一步是:理解业务。
当然了,在真正的数据应用领域,比如金融,医疗,电商,我们的数据不可能像泰坦尼克号数据的特征这样少,这
样明显,那如果遇见极端情况,我们无法依赖对业务的理解来选择特征,该怎么办呢?我们有四种方法可以用来选
择特征:过滤法,嵌入法,包装法,和降维算法 。
#导入数据,让我们使用digit recognizor数据来一展身手
import pandas as pd
data = pd.read_csv(r"C:\work\learnbetter\micro-class\week 3 Preprocessing\digit recognizor.csv")
X = data.iloc[:,1:]
y = data.iloc[:,0]
X.shape
"""
这个数据量相对夸张,如果使用支持向量机和神经网络,很可能会直接跑不出来。使用KNN跑一次大概需要半个小时。用这个数据举例,能更够体现特征工程的重要性。
"""
3.1 Filter过滤法
过滤方法通常用作预处理步骤,特征选择完全独立于任何机器学习算法。它是根据各种统计检验中的分数以及相关
性的各项指标来选择特征。

3.1.1 方差过滤
3.1.1.1 VarianceThreshold
是通过特征本身的方差来筛选特征的类。比如一个特征本身的方差很小,就表示样本在这个特征上基本没有差
异,可能特征中的大多数值都一样,甚至整个特征的取值都相同,那这个特征对于样本区分没有什么作用。所以无
论接下来的特征工程要做什么,都要优先消除方差为0的特征。VarianceThreshold有重要参数threshold,表示方
差的阈值,表示舍弃所有方差小于threshold的特征,不填默认为0,即删除所有的记录都相同的特征。
from sklearn.feature_selection import VarianceThreshold
selector = VarianceThreshold() #实例化,不填参数默认方差为0
X_var0 = selector.fit_transform(X) #获取删除不合格特征之后的新特征矩阵
#也可以直接写成 X = VairanceThreshold().fit_transform(X)
X_var0.shape#(42000, 708)
pd.DataFrame(X_var0).head()可以看见,我们已经删除了方差为0的特征,但是依然剩下了708多个特征,明显还需要进一步的特征选择。然
而,如果我们知道我们需要多少个特征,方差也可以帮助我们将特征选择一步到位。比如说,我们希望留下一半的
特征,那可以设定一个让特征总数减半的方差阈值,只要找到特征方差的中位数,再将这个中位数作为参数
threshold的值输入就好了:
import numpy as np
# X.var()#每一列的方差
X_fsvar = VarianceThreshold(np.median(X.var().values)).fit_transform(X)
X.var().values
np.median(X.var().values)
X_fsvar.shape#(42000, 392)当特征是二分类时,特征的取值就是伯努利随机变量,这些变量的方差可以计算为:
$$Var[X]=p(1-p)$$
其中X是特征矩阵,p是二分类特征中的一类在这个特征中所占的概率。
#若特征是伯努利随机变量,假设p=0.8,即二分类特征中某种分类占到80%以上的时候删除特征
X_bvar = VarianceThreshold(.8 * (1 - .8)).fit_transform(X)
X_bvar.shape3.1.1.2 方差过滤对模型的影响
我们这样做了以后,对模型效果会有怎样的影响呢?在这里,我为大家准备了KNN和随机森林分别在方差过滤前和
方差过滤后运行的效果和运行时间的对比。KNN是K近邻算法中的分类算法,其原理非常简单,是利用每个样本到
其他样本点的距离来判断每个样本点的相似度,然后对样本进行分类。KNN必须遍历每个特征和每个样本,因而特
征越多,KNN的计算也就会越缓慢。由于这一段代码对比运行时间过长,所以我为大家贴出了代码和结果
- 导入模块并准备数据
#KNN vs 随机森林在不同方差过滤效果下的对比
from sklearn.ensemble import RandomForestClassifier as RFC
from sklearn.neighbors import KNeighborsClassifier as KNN
from sklearn.model_selection import cross_val_score
import numpy as np
X = data.iloc[:,1:]
y = data.iloc[:,0]
X_fsvar = VarianceThreshold(np.median(X.var().values)).fit_transform(X)我们从模块neighbors导入KNeighborsClassfier缩写为KNN,导入随机森林缩写为RFC,然后导入交叉验证模块和
numpy。其中未过滤的数据是X和y,使用中位数过滤后的数据是X_fsvar,都是我们之前已经运行过的代码 。
- KNN方差过滤前
#======【TIME WARNING:35mins +】======#
cross_val_score(KNN(),X,y,cv=5).mean()
#python中的魔法命令,可以直接使用%%timeit来计算运行这个cell中的代码所需的时间
#为了计算所需的时间,需要将这个cell中的代码运行很多次(通常是7次)后求平均值,因此运行%%timeit的时间会
# 远远超过cell中的代码单独运行的时间
#======【TIME WARNING:4 hours】======#
%%timeit
cross_val_score(KNN(),X,y,cv=5).mean()

- KNN方差过滤后
#======【TIME WARNING:20 mins+】======#
cross_val_score(KNN(),X_fsvar,y,cv=5).mean()
#======【TIME WARNING:2 hours】======#
%%timeit
cross_val_score(KNN(),X,y,cv=5).mean()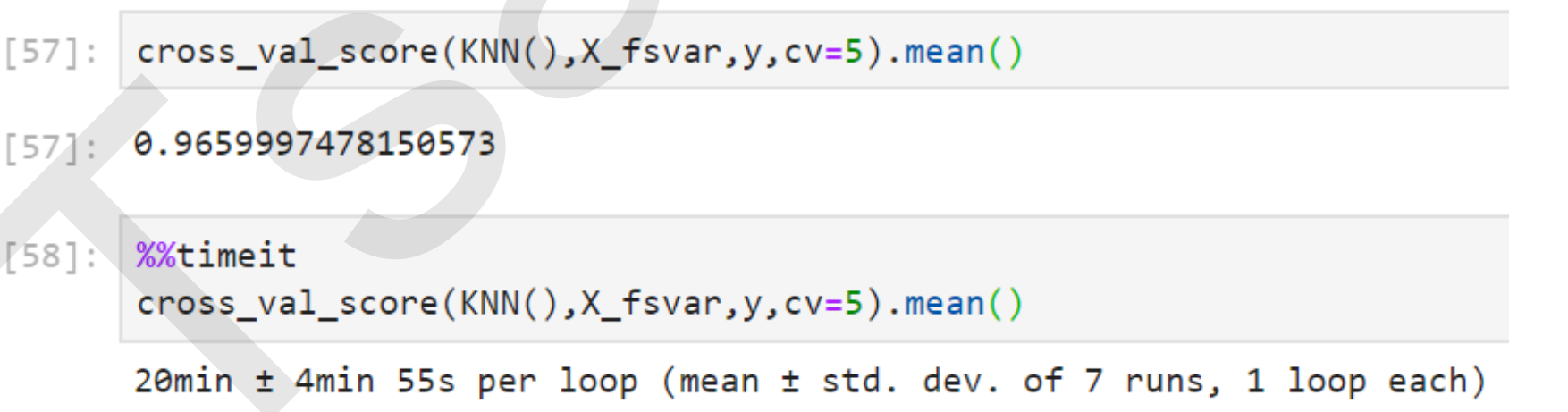
可以看出,对于KNN,过滤后的效果十分明显:准确率稍有提升,但平均运行时间减少了10分钟,特征选择过后算
法的效率上升了1/3。那随机森林又如何呢?
- 随机森林方差过滤前
cross_val_score(RFC(n_estimators=10,random_state=0),X,y,cv=5).mean()
- 随机森林方差过滤后
cross_val_score(RFC(n_estimators=10,random_state=0),X_fsvar,y,cv=5).mean()
首先可以观察到的是,随机森林的准确率略逊于KNN,但运行时间却连KNN的1%都不到,只需要十几秒钟。其
次,方差过滤后,随机森林的准确率也微弱上升,但运行时间却几乎是没什么变化,依然是11秒钟。
为什么随机森林运行如此之快?为什么方差过滤对随机森林没很大的有影响?这是由于两种算法的原理中涉及到的
计算量不同。最近邻算法KNN,单棵决策树,支持向量机SVM,神经网络,回归算法,都需要遍历特征或升维来进
行运算,所以他们本身的运算量就很大,需要的时间就很长,因此方差过滤这样的特征选择对他们来说就尤为重
要。但对于不需要遍历特征的算法,比如随机森林,它随机选取特征进行分枝,本身运算就非常快速,因此特征选
择对它来说效果平平。这其实很容易理解,无论过滤法如何降低特征的数量,随机森林也只会选取固定数量的特征
来建模;而最近邻算法就不同了,特征越少,距离计算的维度就越少,模型明显会随着特征的减少变得轻量。因
此,过滤法的主要对象是:需要遍历特征或升维的算法们,而过滤法的主要目的是:在维持算法表现的前提下,帮
助算法们降低计算成本 。
思考:过滤法对随机森林无效,却对树模型有效?
从算法原理上来说,传统决策树需要遍历所有特征,计算不纯度后进行分枝,而随机森林却是随机选择特征进 行计算和分枝,因此随机森林的运算更快,过滤法对随机森林无用,对决策树却有用
在sklearn中,决策树和随机森林都是随机选择特征进行分枝(不记得的小伙伴可以去复习第一章:决策树, 参数random_state),但决策树在建模过程中随机抽取的特征数目却远远超过随机森林当中每棵树随机抽取 的特征数目(比如说对于这个780维的数据,随机森林每棵树只会抽取10
20个特征,而决策树可能会抽取 300400个特征),因此,过滤法对随机森林无用,却对决策树有用也因此,在sklearn中,随机森林中的每棵树都比单独的一棵决策树简单得多,高维数据下的随机森林的计算 比决策树快很多。
对受影响的算法来说,我们可以将方差过滤的影响总结如下:

在我们的对比当中,我们使用的方差阈值是特征方差的中位数,因此属于阈值比较大,过滤掉的特征比较多的情
况。我们可以观察到,无论是KNN还是随机森林,在过滤掉一半特征之后,模型的精确度都上升了。这说明被我们
过滤掉的特征在当前随机模式(random_state = 0)下大部分是噪音。那我们就可以保留这个去掉了一半特征的数
据,来为之后的特征选择做准备。当然,如果过滤之后模型的效果反而变差了,我们就可以认为,被我们过滤掉的
特征中有很多都有有效特征,那我们就放弃过滤,使用其他手段来进行特征选择。
思考:虽然随机森林算得快,但KNN的效果比随机森林更好?
调整一下n_estimators试试看吧O(∩_∩)O,随机森林是个非常强大的模型哦~
3.1.1.3 选取超参数threshold
我们怎样知道,方差过滤掉的到底时噪音还是有效特征呢?过滤后模型到底会变好还是会变坏呢?答案是:每个数
据集不一样,只能自己去尝试。这里的方差阈值,其实相当于是一个超参数,要选定最优的超参数,我们可以画学
习曲线,找模型效果最好的点。但现实中,我们往往不会这样去做,因为这样会耗费大量的时间。我们只会使用阈
值为0或者阈值很小的方差过滤,来为我们优先消除一些明显用不到的特征,然后我们会选择更优的特征选择方法
继续削减特征数量。
3.1.2 相关性过滤
方差挑选完毕之后,我们就要考虑下一个问题:相关性了。我们希望选出与标签相关且有意义的特征,因为这样的
特征能够为我们提供大量信息。如果特征与标签无关,那只会白白浪费我们的计算内存,可能还会给模型带来噪
音。在sklearn当中,我们有三种常用的方法来评判特征与标签之间的相关性:卡方,F检验,互信息。
3.1.2.1 卡方过滤
卡方过滤是专门针对离散型标签(即分类问题)的相关性过滤。卡方检验类feature_selection.chi2计算每个==非负
特征和标签==(数据预处理)之间的卡方统计量,并依照卡方统计量由高到低为特征排名。再结合feature_selection.SelectKBest
这个可以输入”评分标准“来选出前K个分数最高的特征的类,我们可以借此除去最可能独立于标签,与我们分类目
的无关的特征。
另外,如果卡方检验检测到某个特征中所有的值都相同,会提示我们使用方差先进行方差过滤。并且,刚才我们已
经验证过,当我们使用方差过滤筛选掉一半的特征后,模型的表现时提升的。因此在这里,我们使用threshold=中
位数时完成的方差过滤的数据来做卡方检验(如果方差过滤后模型的表现反而降低了,那我们就不会使用方差过滤
后的数据,而是使用原数据):
from sklearn.ensemble import RandomForestClassifier as RFC
from sklearn.model_selection import cross_val_score
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import chi2
#假设在这里我一直我需要300个特征
X_fschi = SelectKBest(chi2, k=300).fit_transform(X_fsvar, y)
X_fschi.shape验证一下模型的效果如何:
cross_val_score(RFC(n_estimators=10,random_state=0),X_fschi,y,cv=5).mean()可以看出,模型的效果降低了,这说明我们在设定k=300的时候删除了与模型相关且有效的特征,我们的K值设置
得太小,要么我们需要调整K值,要么我们必须放弃相关性过滤。当然,如果模型的表现提升,则说明我们的相关
性过滤是有效的,是过滤掉了模型的噪音的,这时候我们就保留相关性过滤的结果。
3.1.2.2 选取超参数K
那如何设置一个最佳的K值呢?在现实数据中,数据量很大,模型很复杂的时候,我们也许不能先去跑一遍模型看
看效果,而是希望最开始就能够选择一个最优的超参数k。那第一个方法,就是我们之前提过的学习曲线:
#======【TIME WARNING: 5 mins】======#
%matplotlib inline
import matplotlib.pyplot as plt
score = []
for i in range(390,200,-10):
X_fschi = SelectKBest(chi2, k=i).fit_transform(X_fsvar, y)
once = cross_val_score(RFC(n_estimators=10,random_state=0),X_fschi,y,cv=5).mean()
score.append(once)
plt.plot(range(350,200,-10),score)
plt.show()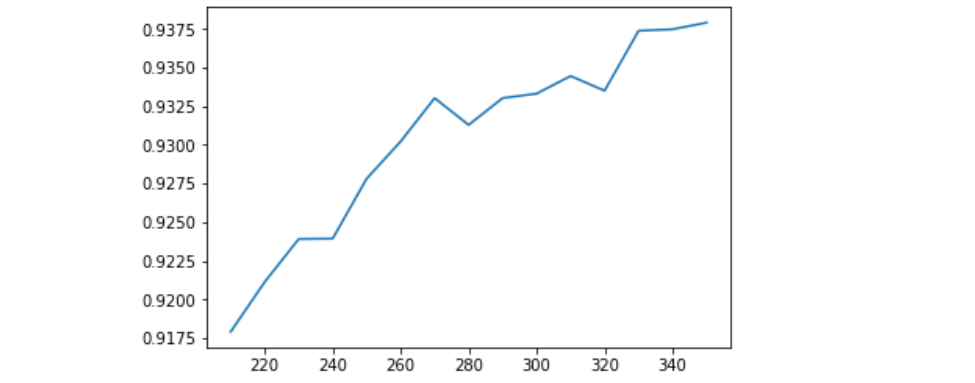
通过这条曲线,我们可以观察到,随着K值的不断增加,模型的表现不断上升,这说明,K越大越好,数据中所有的
特征都是与标签相关的。但是运行这条曲线的时间同样也是非常地长,接下来我们就来介绍一种更好的选择k的方
法:看p值选择k。
卡方检验的本质是推测两组数据之间的差异,其检验的原假设是”两组数据是相互独立的”。卡方检验返回卡方值和
P值两个统计量,其中卡方值很难界定有效的范围,而p值,我们一般使用0.01或0.05作为显著性水平,即p值判断
的边界,具体我们可以这样来看:
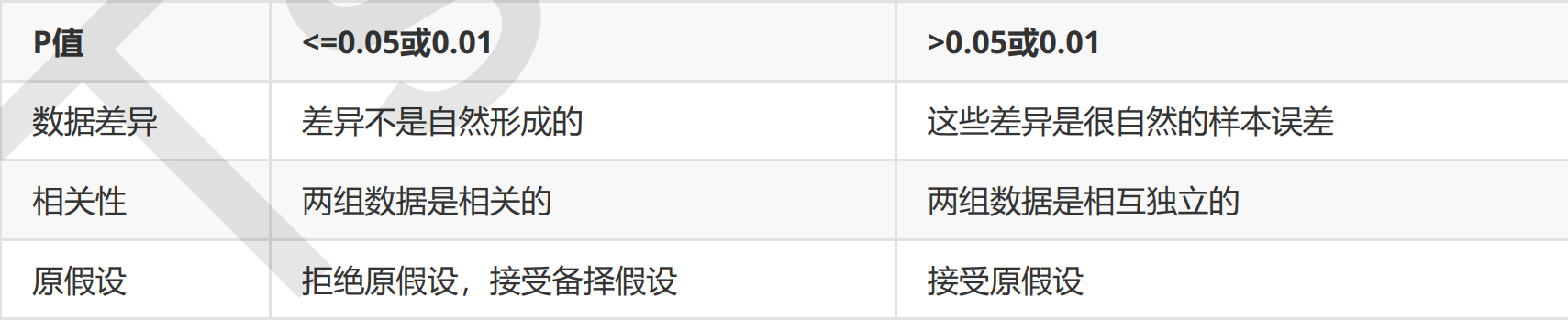
从特征工程的角度,我们希望选取卡方值很大,p值小于0.05的特征,即和标签是相关联的特征。而调用
SelectKBest之前,我们可以直接从chi2实例化后的模型中获得各个特征所对应的卡方值和P值
chivalue, pvalues_chi = chi2(X_fsvar,y)
chivalue
pvalues_chi
#k取多少?我们想要消除所有p值大于设定值,比如0.05或0.01的特征:
k = chivalue.shape[0] - (pvalues_chi > 0.05).sum()
#X_fschi = SelectKBest(chi2, k=填写具体的k).fit_transform(X_fsvar, y)
#cross_val_score(RFC(n_estimators=10,random_state=0),X_fschi,y,cv=5).mean()可以观察到,所有特征的p值都是0,这说明对于digit recognizor这个数据集来说,方差验证已经把所有和标签无
关的特征都剔除了,或者这个数据集本身就不含与标签无关的特征。在这种情况下,舍弃任何一个特征,都会舍弃
对模型有用的信息,而使模型表现下降,因此在我们对计算速度感到满意时,我们不需要使用相关性过滤来过滤我
们的数据。如果我们认为运算速度太缓慢,那我们可以酌情删除一些特征,但前提是,我们必须牺牲模型的表现。
接下来,我们试试看用其他的相关性过滤方法验证一下我们在这个数据集上的结论。
3.1.2.3 F检验
F检验,又称ANOVA,方差齐性检验,是用来捕捉每个特征与标签之间的==线性关系==的过滤方法。它即可以做回归也
可以做分类,因此包含feature_selection.f_classif(F检验分类)和feature_selection.f_regression(F检验回
归)两个类。其中F检验分类用于标签是离散型变量的数据,而F检验回归用于标签是连续型变量的数据。
和卡方检验一样,这两个类需要和类SelectKBest连用,并且我们也可以直接通过输出的统计量来判断我们到底要
设置一个什么样的K。需要注意的是,F检验在数据服从正态分布时效果会非常稳定,因此如果使用F检验过滤,我
们会先将数据转换成服从正态分布的方式。
F检验的本质是寻找两组数据之间的线性关系,其原假设是”数据不存在显著的线性关系“。它返回F值和p值两个统
计量。和卡方过滤一样,我们希望选取p值小于0.05或0.01的特征,这些特征与标签时显著线性相关的,而p值大于
0.05或0.01的特征则被我们认为是和标签没有显著线性关系的特征,应该被删除。以F检验的分类为例,我们继续
在数字数据集上来进行特征选择:
rom sklearn.feature_selection import f_classif
F, pvalues_f = f_classif(X_fsvar,y)
F
pvalues_f
k = F.shape[0] - (pvalues_f > 0.05).sum()
#X_fsF = SelectKBest(f_classif, k=填写具体的k).fit_transform(X_fsvar, y)
#cross_val_score(RFC(n_estimators=10,random_state=0),X_fsF,y,cv=5).mean()得到的结论和我们用卡方过滤得到的结论一模一样:没有任何特征的p值大于0.01,所有的特征都是和标签相关
的,因此我们不需要相关性过滤。
3.1.2.4 互信息法
互信息法是用来捕捉每个特征与标签之间的==任意关系==(包括线性和非线性关系)的过滤方法。和F检验相似,它既
可以做回归也可以做分类,并且包含两个类feature_selection.mutual_info_classif(互信息分类)和
feature_selection.mutual_info_regression(互信息回归)。这两个类的用法和参数都和F检验一模一样,不过
互信息法比F检验更加强大,F检验只能够找出线性关系,而互信息法可以找出任意关系。
互信息法不返回p值或F值类似的统计量,它返回“每个特征与目标之间的互信息量的估计”,这个估计量在[0,1]之间
取值,为0则表示两个变量独立,为1则表示两个变量完全相关。以互信息分类为例的代码如下
from sklearn.feature_selection import mutual_info_classif as MIC
result = MIC(X_fsvar,y)
k = result.shape[0] - sum(result <= 0)
#X_fsmic = SelectKBest(MIC, k=填写具体的k).fit_transform(X_fsvar, y)
#cross_val_score(RFC(n_estimators=10,random_state=0),X_fsmic,y,cv=5).mean()所有特征的互信息量估计都大于0,因此所有特征都与标签相关。
当然了,无论是F检验还是互信息法,大家也都可以使用学习曲线,只是使用统计量的方法会更加高效。当统计量
判断已经没有特征可以删除时,无论用学习曲线如何跑,删除特征都只会降低模型的表现。当然了,如果数据量太
庞大,模型太复杂,我们还是可以牺牲模型表现来提升模型速度,一切都看大家的具体需求。
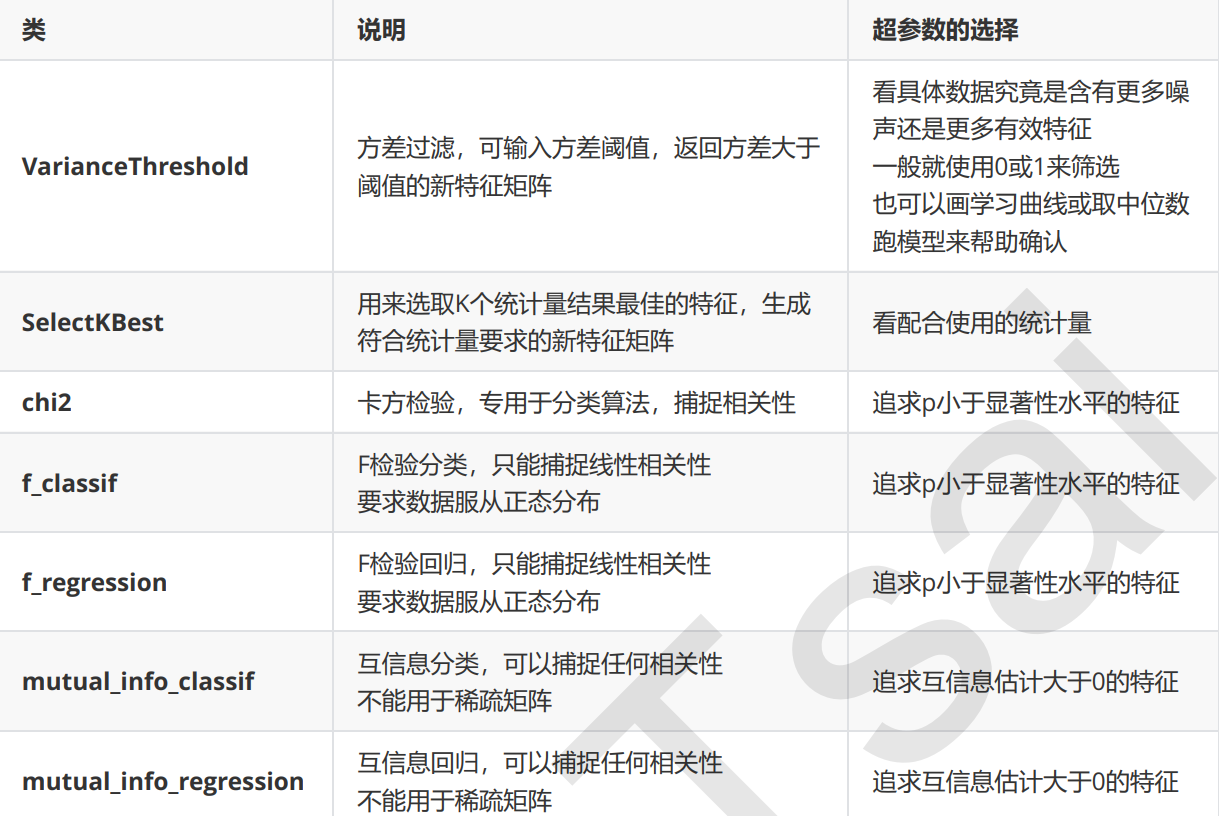
3.1.3 过滤法总结
到这里我们学习了常用的基于过滤法的特征选择,包括方差过滤,基于卡方,F检验和互信息的相关性过滤,讲解
了各个过滤的原理和面临的问题,以及怎样调这些过滤类的超参数。通常来说,我会建议,先使用方差过滤,然后
使用互信息法来捕捉相关性,不过了解各种各样的过滤方式也是必要的。所有信息被总结在下表,大家自取
3.2 Embedded嵌入法
嵌入法是一种让算法自己决定使用哪些特征的方法,即特征选择和算法训练同时进行。在使用嵌入法时,我们先使
用某些机器学习的算法和模型进行训练,得到各个特征的权值系数,根据权值系数从大到小选择特征。这些权值系
数往往代表了特征对于模型的某种贡献或某种重要性,比如决策树和树的集成模型中的feature_importances_属
性,可以列出各个特征对树的建立的贡献,我们就可以基于这种贡献的评估,找出对模型建立最有用的特征。因此
相比于过滤法,嵌入法的结果会更加精确到模型的效用本身,对于提高模型效力有更好的效果。并且,由于考虑特
征对模型的贡献,因此无关的特征(需要相关性过滤的特征)和无区分度的特征(需要方差过滤的特征)都会因为
缺乏对模型的贡献而被删除掉,可谓是过滤法的进化版。
然而,嵌入法也不是没有缺点。
过滤法中使用的统计量可以使用统计知识和常识来查找范围(如p值应当低于显著性水平0.05),而嵌入法中使用
的权值系数却没有这样的范围可找——我们可以说,权值系数为0的特征对模型丝毫没有作用,但当大量特征都对
模型有贡献且贡献不一时,我们就很难去界定一个有效的临界值。这种情况下,模型权值系数就是我们的超参数,
我们或许需要学习曲线,或者根据模型本身的某些性质去判断这个超参数的最佳值究竟应该是多少。在我们之后的
学习当中,每次讲解新的算法,我都会为大家提到这个算法中的特征工程是如何处理,包括具体到每个算法的嵌入
法如何使用。在这堂课中,我们会为大家讲解随机森林和决策树模型的嵌入法。
另外,嵌入法引入了算法来挑选特征,因此其计算速度也会和应用的算法有很大的关系。如果采用计算量很大,计
算缓慢的算法,嵌入法本身也会非常耗时耗力。并且,在选择完毕之后,我们还是需要自己来评估模型。
- feature_selection.SelectFromModel
class sklearn.feature_selection.SelectFromModel (estimator, threshold=None, prefit=False, norm_order=1,max_features=None)
SelectFromModel是一个元变换器,可以与任何在拟合后具有coef_,feature_importances_属性或参数中可选惩
罚项的评估器一起使用(比如随机森林和树模型就具有属性feature_importances_,逻辑回归就带有l1和l2惩罚
项,线性支持向量机也支持l2惩罚项)。
对于有feature_importances_的模型来说,若重要性低于提供的阈值参数,则认为这些特征不重要并被移除。
feature_importances_的取值范围是[0,1],如果设置阈值很小,比如0.001,就可以删除那些对标签预测完全没贡
献的特征。如果设置得很接近1,可能只有一两个特征能够被留下。
选读:使用惩罚项的模型的嵌入法 而对于使用惩罚项的模型来说,正则化惩罚项越大,特征在模型中对应的系数就会越小。当正则化惩罚项大到 一定的程度的时候,部分特征系数会变成0,当正则化惩罚项继续增大到一定程度时,所有的特征系数都会趋 于0。 但是我们会发现一部分特征系数会更容易先变成0,这部分系数就是可以筛掉的。也就是说,我们选择 特征系数较大的特征。另外,支持向量机和逻辑回归使用参数C来控制返回的特征矩阵的稀疏性,参数C越 小,返回的特征越少。Lasso回归,用alpha参数来控制返回的特征矩阵,alpha的值越大,返回的特征越少。
| 参数 | 说明 |
|---|---|
| estimator | 使用的模型评估器,只要是带feature_importances_或者coef_属性,或带有l1和l2惩罚 项的模型都可以使用 |
| threshold | 特征重要性的阈值,重要性低于这个阈值的特征都将被删除 |
| prefit | 默认False,判断是否将实例化后的模型直接传递给构造函数。如果为True,则必须直接 调用fit和transform,不能使用fit_transform,并且SelectFromModel不能与 cross_val_score,GridSearchCV和克隆估计器的类似实用程序一起使用。 |
| norm_order | k可输入非零整数,正无穷,负无穷,默认值为1 在评估器的coef_属性高于一维的情况下,用于过滤低于阈值的系数的向量的范数的阶 数。 |
| max_features | 在阈值设定下,要选择的最大特征数。要禁用阈值并仅根据max_features选择,请设置 threshold = -np.inf |
我们重点要考虑的是前两个参数。在这里,我们使用随机森林为例,则需要学习曲线来帮助我们寻找最佳特征值。
from sklearn.feature_selection import SelectFromModel
from sklearn.ensemble import RandomForestClassifier as RFC
RFC_ = RFC(n_estimators =10,random_state=0)
X_embedded = SelectFromModel(RFC_,threshold=0.005).fit_transform(X,y)
#在这里我只想取出来有限的特征。0.005这个阈值对于有780个特征的数据来说,是非常高的阈值,因为平均每个特征只能够分到大约0.001的feature_importances_
X_embedded.shape
#模型的维度明显被降低了
#同样的,我们也可以画学习曲线来找最佳阈值
#======【TIME WARNING:10 mins】======#
import numpy as np
import matplotlib.pyplot as plt
RFC_.fit(X,y).feature_importances_
threshold = np.linspace(0,(RFC_.fit(X,y).feature_importances_).max(),20)
score = []
for i in threshold:
X_embedded = SelectFromModel(RFC_,threshold=i).fit_transform(X,y)
once = cross_val_score(RFC_,X_embedded,y,cv=5).mean()
score.append(once)
plt.plot(threshold,score)
plt.show()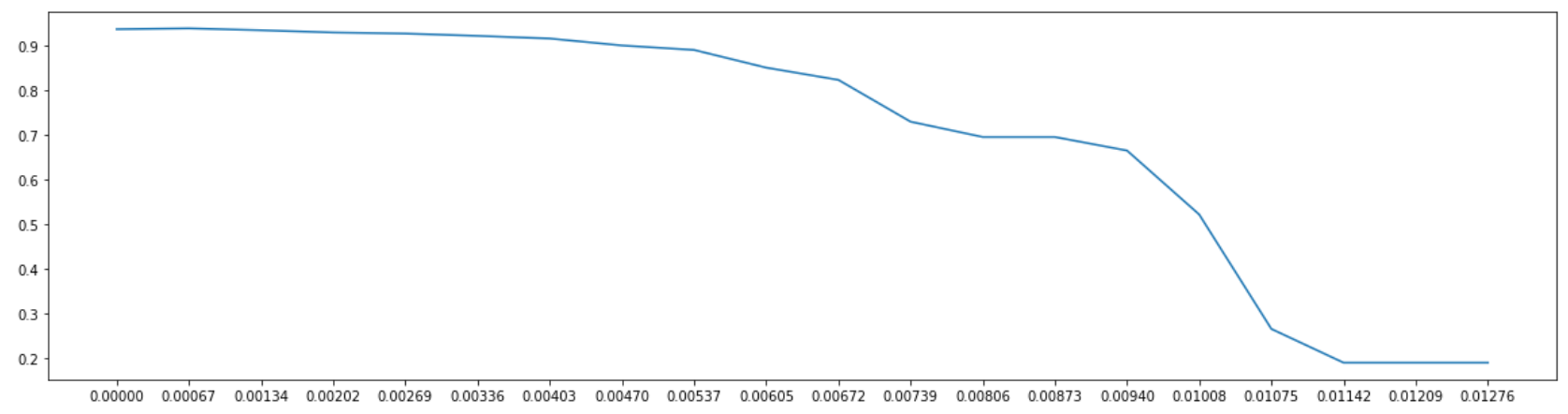
从图像上来看,随着阈值越来越高,模型的效果逐渐变差,被删除的特征越来越多,信息损失也逐渐变大。但是在
0.00134之前,模型的效果都可以维持在0.93以上,因此我们可以从中挑选一个数值来验证一下模型的效果。
X_embedded = SelectFromModel(RFC_,threshold=0.00067).fit_transform(X,y)
X_embedded.shape
cross_val_score(RFC_,X_embedded,y,cv=5).mean()可以看出,特征个数瞬间缩小到324多,这比我们在方差过滤的时候选择中位数过滤出来的结果392列要小,并且
交叉验证分数0.9399高于方差过滤后的结果0.9388,这是由于嵌入法比方差过滤更具体到模型的表现的缘故,换一
个算法,使用同样的阈值,效果可能就没有这么好了。
和其他调参一样,我们可以在第一条学习曲线后选定一个范围,使用细化的学习曲线来找到最佳值:
#======【TIME WARNING:10 mins】======#
score2 = []
for i in np.linspace(0,0.00134,20):
X_embedded = SelectFromModel(RFC_,threshold=i).fit_transform(X,y)
once = cross_val_score(RFC_,X_embedded,y,cv=5).mean()
score2.append(once)
plt.figure(figsize=[20,5])
plt.plot(np.linspace(0,0.00134,20),score2)
plt.xticks(np.linspace(0,0.00134,20))
plt.show()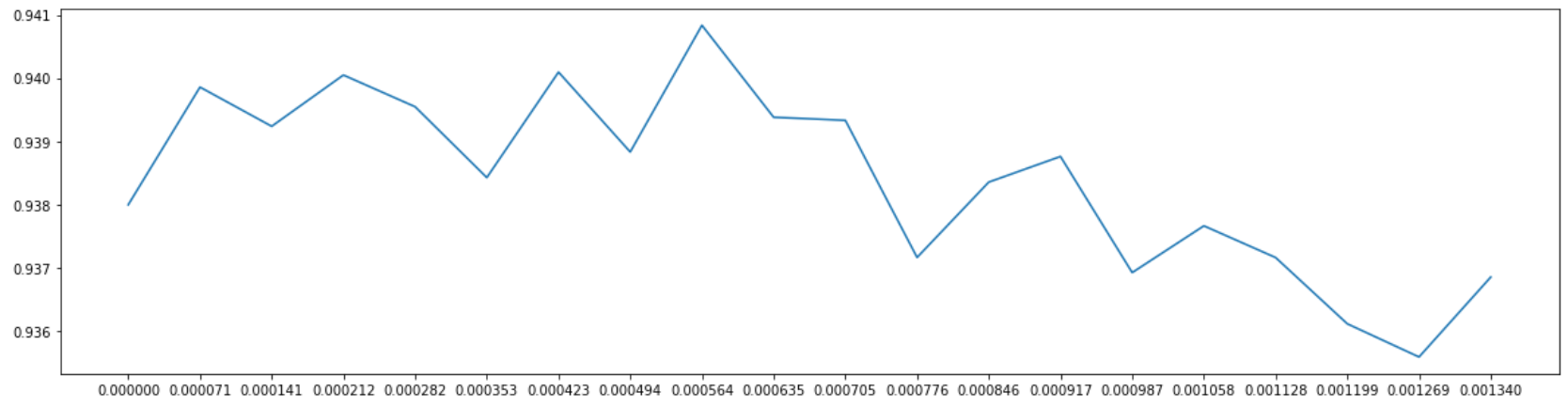
查看结果,果然0.00067并不是最高点,真正的最高点0.000564已经将模型效果提升到了94%以上。我们使用
0.000564来跑一跑我们的SelectFromModel
X_embedded = SelectFromModel(RFC_,threshold=0.000564).fit_transform(X,y)
X_embedded.shape
cross_val_score(RFC_,X_embedded,y,cv=5).mean()
#=====【TIME WARNING:2 min】=====#
#我们可能已经找到了现有模型下的最佳结果,如果我们调整一下随机森林的参数呢?
cross_val_score(RFC(n_estimators=100,random_state=0),X_embedded,y,cv=5).mean()得出的特征数目依然小于方差筛选,并且模型的表现也比没有筛选之前更高,已经完全可以和计算一次半小时的
KNN相匹敌(KNN的准确率是96.58%),接下来再对随机森林进行调参,准确率应该还可以再升高不少。可见,
在嵌入法下,我们很容易就能够实现特征选择的目标:减少计算量,提升模型表现。因此,比起要思考很多统计量
的过滤法来说,嵌入法可能是更有效的一种方法。然而,在算法本身很复杂的时候,过滤法的计算远远比嵌入法要
快,所以大型数据中,我们还是会优先考虑过滤法。
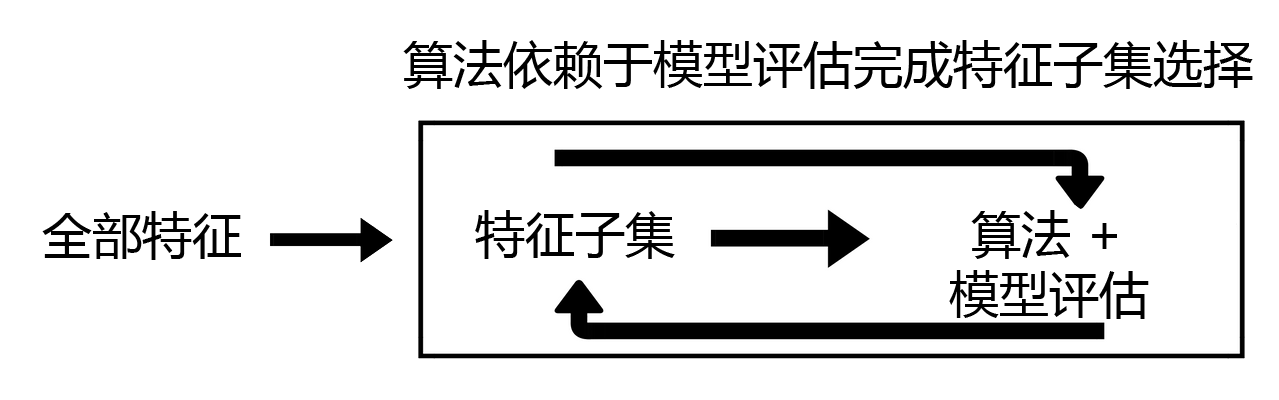
3.3 Wrapper包装法
包装法也是一个特征选择和算法训练同时进行的方法,与嵌入法十分相似,它也是依赖于算法自身的选择,比如
coef_属性或feature_importances_属性来完成特征选择。但不同的是,我们往往使用一个目标函数作为黑盒来帮
助我们选取特征,而不是自己输入某个评估指标或统计量的阈值。包装法在初始特征集上训练评估器,并且通过
coef_属性或通过feature_importances_属性获得每个特征的重要性。然后,从当前的一组特征中修剪最不重要的
特征。在修剪的集合上递归地重复该过程,直到最终到达所需数量的要选择的特征。区别于过滤法和嵌入法的一次
训练解决所有问题,包装法要使用特征子集进行多次训练,因此它所需要的计算成本是最高的。
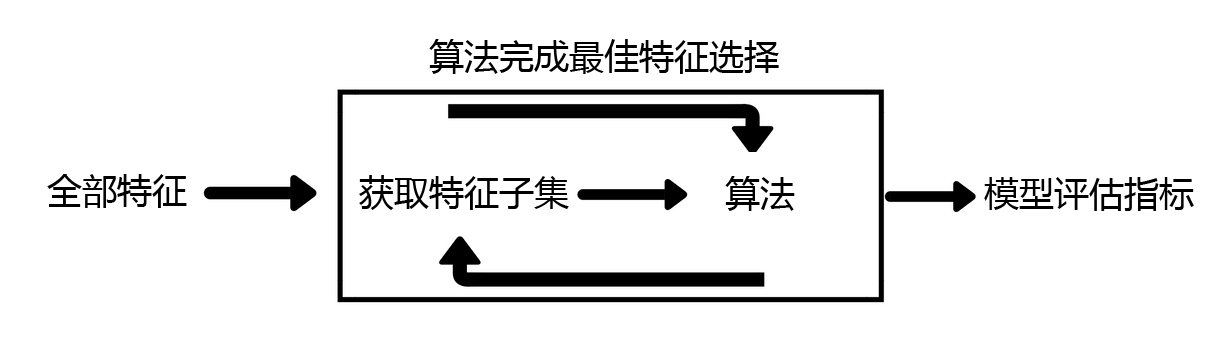
注意,在这个图中的“算法”,指的不是我们最终用来导入数据的分类或回归算法(即不是随机森林),而是专业的
数据挖掘算法,即我们的目标函数。这些数据挖掘算法的核心功能就是选取最佳特征子集。
最典型的目标函数是递归特征消除法(Recursive feature elimination, 简写为RFE)。它是一种贪婪的优化算法,
旨在找到性能最佳的特征子集。 它反复创建模型,并在每次迭代时保留最佳特征或剔除最差特征,下一次迭代时,
它会使用上一次建模中没有被选中的特征来构建下一个模型,直到所有特征都耗尽为止。 然后,它根据自己保留或
剔除特征的顺序来对特征进行排名,最终选出一个最佳子集。包装法的效果是所有特征选择方法中最利于提升模型
表现的,它可以使用很少的特征达到很优秀的效果。除此之外,在特征数目相同时,包装法和嵌入法的效果能够匹
敌,不过它比嵌入法算得更见缓慢,所以也不适用于太大型的数据。相比之下,包装法是最能保证模型效果的特征
选择方法
- feature_selection.RFE
class sklearn.feature_selection.RFE (estimator, n_features_to_select=None, step=1, verbose=0)
参数estimator是需要填写的实例化后的评估器,n_features_to_select是想要选择的特征个数,step表示每次迭
代中希望移除的特征个数。除此之外,RFE类有两个很重要的属性,.support_:返回所有的特征的是否最后被选
中的布尔矩阵,以及.ranking_返回特征的按数次迭代中综合重要性的排名。类feature_selection.RFECV会在交叉
验证循环中执行RFE以找到最佳数量的特征,增加参数cv,其他用法都和RFE一模一样。
from sklearn.feature_selection import RFE
RFC_ = RFC(n_estimators =10,random_state=0)
selector = RFE(RFC_, n_features_to_select=340, step=50).fit(X, y)
selector.support_.sum()
selector.ranking_
X_wrapper = selector.transform(X)
cross_val_score(RFC_,X_wrapper,y,cv=5).mean()我们也可以对包装法画学习曲线:
#======【TIME WARNING: 15 mins】======#
score = []
for i in range(1,751,50):
X_wrapper = RFE(RFC_,n_features_to_select=i, step=50).fit_transform(X,y)
once = cross_val_score(RFC_,X_wrapper,y,cv=5).mean()
score.append(once)
plt.figure(figsize=[20,5])
plt.plot(range(1,751,50),score)
plt.xticks(range(1,751,50))
plt.show()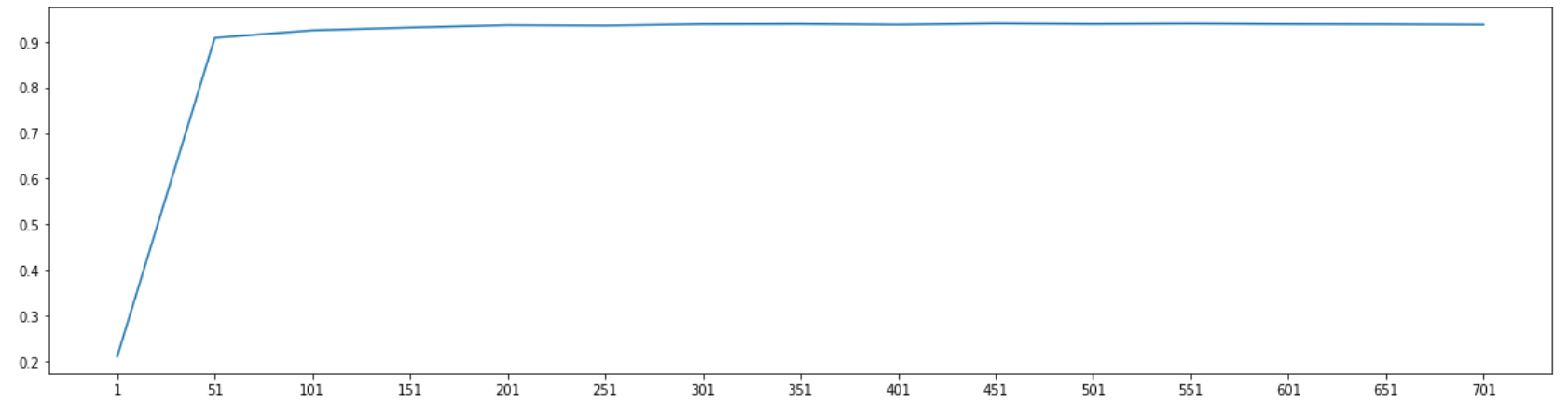
明显能够看出,在包装法下面,应用50个特征时,模型的表现就已经达到了90%以上,比嵌入法和过滤法都高效很
多。我们可以放大图像,寻找模型变得非常稳定的点来画进一步的学习曲线(就像我们在嵌入法中做的那样)。如
果我们此时追求的是最大化降低模型的运行时间,我们甚至可以直接选择50作为特征的数目,这是一个在缩减了
94%的特征的基础上,还能保证模型表现在90%以上的特征组合,不可谓不高效。
同时,我们提到过,在特征数目相同时,包装法能够在效果上匹敌嵌入法。试试看如果我们也使用340作为特征数
目,运行一下,可以感受一下包装法和嵌入法哪一个的速度更加快。由于包装法效果和嵌入法相差不多,在更小的
范围内使用学习曲线,我们也可以将包装法的效果调得很好,大家可以去试试看。
3.4 特征选择总结
至此,我们讲完了降维之外的所有特征选择的方法。这些方法的代码都不难,但是每种方法的原理都不同,并且都
涉及到不同调整方法的超参数。经验来说,过滤法更快速,但更粗糙。包装法和嵌入法更精确,比较适合具体到算
法去调整,但计算量比较大,运行时间长。当数据量很大的时候,优先使用方差过滤和互信息法调整,再上其他特
征选择方法。使用逻辑回归时,优先使用嵌入法。使用支持向量机时,优先使用包装法。迷茫的时候,从过滤法走
起,看具体数据具体分析。
其实特征选择只是特征工程中的第一步。真正的高手,往往使用特征创造或特征提取来寻找高级特征。在Kaggle之
类的算法竞赛中,很多高分团队都是在高级特征上做文章,而这是比调参和特征选择更难的,提升算法表现的高深
方法。特征工程非常深奥,虽然我们日常可能用到不多,但其实它非常美妙。若大家感兴趣,也可以自己去网上搜
一搜,多读多看多试多想,技术逐渐会成为你的囊中之物。
sklearn :Pipeline 与 FeatureUnion入门指南
Pipeline的作用
Pipeline可以将许多算法模型串联起来,可以用于把多个estamitors级联成一个estamitor,比如将特征提取、归一化、分类组织在一起形成一个典型的机器学习问题工作流。Pipleline中最后一个之外的所有estimators都必须是变换器(transformers),最后一个estimator可以是任意类型(transformer,classifier,regresser),若最后一个estimator是个分类器,则整个pipeline就可以作为分类器使用,如果最后一个estimator是个聚类器,则整个pipeline就可以作为聚类器使用。
实际上,调用pipeline的fit方法,是用前n-1个变换器处理特征,之后传递给最后的estimator训练。pipeline继承最后一个estimator的所有方法。
由此带来两点直接好处:
- 1.直接调用fit和predict方法来对pipeline中的所有算法模型进行训练和预测。
- 2.可以结合grid search对参数进行选择。
Pipeline的参数与使用
function
Pipline的方法都是执行各个学习器中对应的方法,如果该学习器没有该方法,会报错 假设该Pipline共有n个学习器。
- transform,依次执行各个学习器的transform方法
- inverse_transform,依次执行各个学习器的inverse_transform方法
- fit,依次对前n-1个学习器执行fit和transform方法,第n个学习器(最后一个学习器)执行fit方法
- predict,执行第n个学习器的predict方法
- score,执行第n个学习器的score方法
- set_params,设置第n个学习器的参数
- get_param,获取第n个学习器的参数
用法(例子)
步骤:1.首先对数据进行预处理,比如缺失值的处理
2.数据的标准化
3.降维
4.特征选择算法
5.分类或者预测或者聚类算法(估计器,estimator)
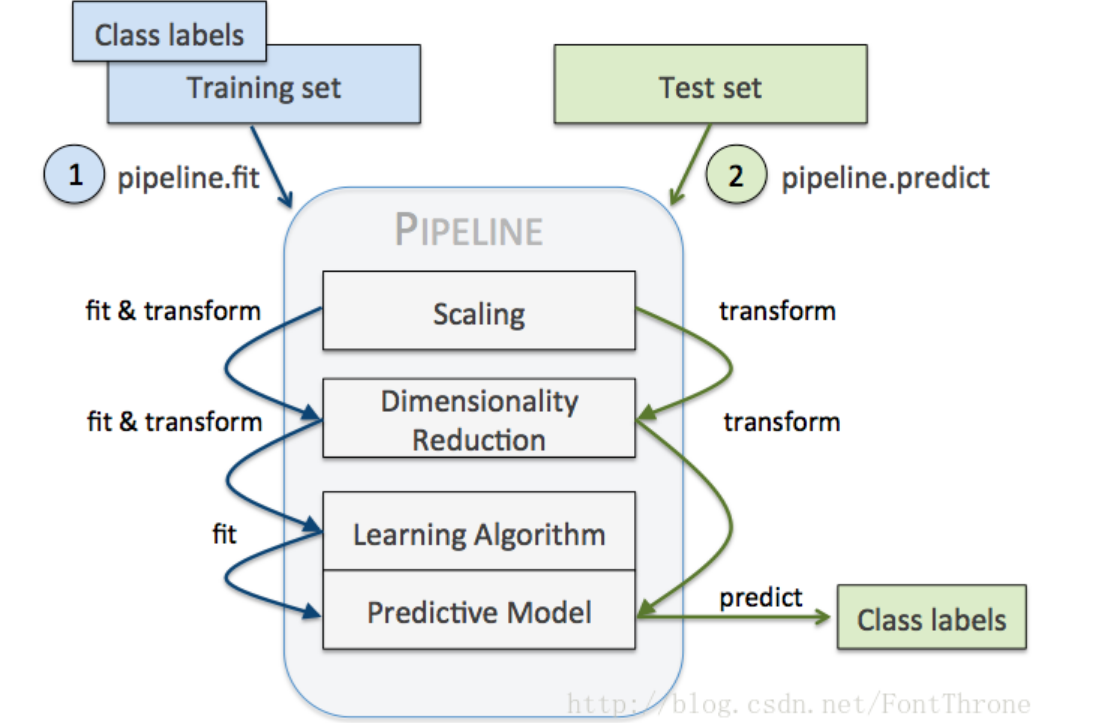
Pipeline的调用
Pipeline是使用(key, value)对的列表构建的,其中key是包含要给予此步骤的名称的字符串,value是对应的不同学习器。
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
iris=load_iris()
pipe=Pipeline([('sc', StandardScaler()),('pca',PCA()),('svc',SVC())])
#例如‘sc’是StandardScaler()的简称,亦或者是代称
pipe.fit(iris.data,iris.target)输出:
Pipeline(memory=None,
steps=[('sc', StandardScaler(copy=True, with_mean=True, with_std=True)), ('pca', PCA(copy=True, iterated_power='auto', n_components=None, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)), ('svc', SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0,
decision_function_shape='ovr', degree=3, gamma='auto_deprecated',
kernel='rbf', max_iter=-1, probability=False, random_state=None,
shrinking=True, tol=0.001, verbose=False))])
其次可以通过make_pipeline函数实现pipeline的调用:它是Pipeline类的简单实现,只需传入每个step的类实例即可,不需自己命名,自动将类的小写设为该step的名:
from sklearn.linear_model import ElasticNet, Lasso, BayesianRidge, LassoLarsIC
from sklearn.pipeline import make_pipeline
from sklearn.preprocessing import RobustScaler #用来解决离群点
new_pipeline=make_pipeline(RobustScaler(), Lasso(alpha =0.0005, random_state=1))输出:
Pipeline(memory=None,
steps=[('robustscaler', RobustScaler(copy=True, quantile_range=(25.0, 75.0), with_centering=True,
with_scaling=True)), ('lasso', Lasso(alpha=0.0005, copy_X=True, fit_intercept=True, max_iter=1000,
normalize=False, positive=False, precompute=False, random_state=1,
selection='cyclic', tol=0.0001, warm_start=False))])Pipeline参数改变
可以通过set_params重新设置每个类里边需传入的参数,设置方法为step的name__parma=参数值:
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
iris=load_iris()
pipe=Pipeline([('sc', StandardScaler()),('pca',PCA()),('svc',SVC())])
#例如‘sc’是StandardScaler()的简称,亦或者是代称
pipe.set_params(sc__copy=False)
#改变参数的格式为 学习器简称__该学习器对应参数名=参数值
pipe.fit(iris.data,iris.target)
输出:
#可以看到sc中的copy确实由true改为false
Pipeline(memory=None,
steps=[('sc', StandardScaler(copy=False, with_mean=True, with_std=True)), ('pca', PCA(copy=True, iterated_power='auto', n_components=None, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)), ('svc', SVC(C=1.0, cache_size=200, class_weight=None, coef0=0.0,
decision_function_shape='ovr', degree=3, gamma='auto_deprecated',
kernel='rbf', max_iter=-1, probability=False, random_state=None,
shrinking=True, tol=0.001, verbose=False))])
Pipeline与GridSearch
举例为pipeline与GridSearch在pca与svc中的使用:
from sklearn.model_selection import GridSearchCV
#以下格式为 学习器简写__该学习器对应的某个参数名=可选参数值范围
params = dict(pca__n_components=[2, 5, 10],
svc__C=[0.1, 10, 100])
grid_search = GridSearchCV(pipe, param_grid=params)
FeatureUnion的作用
FeatureUnion把若干个transformer object组合成一个新的estimators。这个新的transformer组合了他们的输出,一个FeatureUnion对象接受一个transformer对象列表。
在训练阶段,每一个transformer都在数据集上独立的训练。在数据变换阶段,多有的训练好的Trandformer可以并行的执行。他们输出的样本特征向量被以end-to-end的方式拼接成为一个更大的特征向量。
- 你只需要调用一次fit和transform就可以在数据集上训练一组estimators。
- 可以把grid search用在FeatureUnion中所有的estimators的参数这上面。
FeatureUnion和Pipeline可以组合使用来创建更加复杂的模型。
注意:FeatureUnion无法检查两个transformers是否产生了相同的特征输出,它仅仅产生了一个原来互相分离的特征向量的集合。确保其产生不一样的特征输出是调用者的事情。
FeatureUnion的调用
FeatureUnion对象实例使用(key, value)构成的list来构造,key是你自己起的transformation的名称,value是一个estimator对象。
from sklearn.pipeline import FeatureUnion
from sklearn.decomposition import PCA
from sklearn.decomposition import KernelPCA
estimators = [('linear_pca', PCA()), ('kernel_pca', KernelPCA())]
combined = FeatureUnion(estimators)
输出:
FeatureUnion(n_jobs=None,
transformer_list=[('linear_pca', PCA(copy=True, iterated_power='auto', n_components=None, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)), ('kernel_pca', KernelPCA(alpha=1.0, coef0=1, copy_X=True, degree=3, eigen_solver='auto',
fit_inverse_transform=False, gamma=None, kernel='linear',
kernel_params=None, max_iter=None, n_components=None, n_jobs=None,
random_state=None, remove_zero_eig=False, tol=0))],
transformer_weights=None)
FeatureUnion的参数变更
from sklearn.pipeline import FeatureUnion
from sklearn.decomposition import PCA
from sklearn.decomposition import KernelPCA
estimators = [('linear_pca', PCA()), ('kernel', KernelPCA())]
combined = FeatureUnion(estimators)
combined.set_params(kernel__alpha=0.6)##注意这里是两个下划线不然会报错
print(combined)
输出:
FeatureUnion(n_jobs=None,
transformer_list=[('linear_pca', PCA(copy=True, iterated_power='auto', n_components=None, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)), ('kernel', KernelPCA(alpha=0.6, coef0=1, copy_X=True, degree=3, eigen_solver='auto',
fit_inverse_transform=False, gamma=None, kernel='linear',
kernel_params=None, max_iter=None, n_components=None, n_jobs=None,
random_state=None, remove_zero_eig=False, tol=0))],
transformer_weights=None)
FeatureUnion与pipeline的区别
pipeline相当于feature串行处理,后一个transformer处理前一个transformer的feature结果;
featureunion相当于feature的并行处理,将所有transformer的处理结果拼接成大的feature vector。
FeatureUnion与pipeline的结合使用
# Author: Andreas Mueller <amueller@ais.uni-bonn.de>
#
# License: BSD 3 clause
from sklearn.pipeline import Pipeline, FeatureUnion
from sklearn.grid_search import GridSearchCV
from sklearn.svm import SVC
from sklearn.datasets import load_iris
from sklearn.decomposition import PCA
from sklearn.feature_selection import SelectKBest
iris = load_iris()
X, y = iris.data, iris.target
# This dataset is way to high-dimensional. Better do PCA:
pca = PCA(n_components=2)
# Maybe some original features where good, too?
selection = SelectKBest(k=1)
# Build estimator from PCA and Univariate selection:
combined_features = FeatureUnion([("pca", pca), ("univ_select", selection)])
# Use combined features to transform dataset:
X_features = combined_features.fit(X, y).transform(X)
svm = SVC(kernel="linear")
# Do grid search over k, n_components and C:
pipeline = Pipeline([("features", combined_features), ("svm", svm)])
param_grid = dict(features__pca__n_components=[1, 2, 3],
features__univ_select__k=[1, 2],
svm__C=[0.1, 1, 10])
grid_search = GridSearchCV(pipeline, param_grid=param_grid, verbose=10)
grid_search.fit(X, y)
print(grid_search.best_estimator_)
输出:、
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1, score=0.960784 - 0.0s
[Parallel(n_jobs=1)]: Done 1 out of 1 | elapsed: 0.0s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1, score=0.901961 - 0.0s
[Parallel(n_jobs=1)]: Done 2 out of 2 | elapsed: 0.0s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=1, score=0.979167 - 0.0s
[Parallel(n_jobs=1)]: Done 3 out of 3 | elapsed: 0.0s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1, score=0.941176 - 0.0s
[Parallel(n_jobs=1)]: Done 4 out of 4 | elapsed: 0.0s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1, score=0.921569 - 0.0s
[Parallel(n_jobs=1)]: Done 5 out of 5 | elapsed: 0.0s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=1, score=0.979167 - 0.0s
[Parallel(n_jobs=1)]: Done 6 out of 6 | elapsed: 0.1s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1, score=0.960784 - 0.0s
[Parallel(n_jobs=1)]: Done 7 out of 7 | elapsed: 0.1s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1, score=0.921569 - 0.0s
[Parallel(n_jobs=1)]: Done 8 out of 8 | elapsed: 0.1s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=1, score=0.979167 - 0.0s
[Parallel(n_jobs=1)]: Done 9 out of 9 | elapsed: 0.1s remaining: 0.0s
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2, score=0.960784 - 0.0s
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2, score=0.921569 - 0.0s
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=0.1, features__univ_select__k=2, score=0.979167 - 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2, score=0.960784 - 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2, score=0.921569 - 0.0s
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=1, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2, score=0.980392 - 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2, score=0.901961 - 0.0s
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=1, svm__C=10, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1, score=0.960784 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1, score=0.901961 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=1, score=0.979167 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1, score=0.980392 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1, score=0.941176 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=1, score=0.979167 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1, score=0.980392 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1, score=0.941176 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=1, score=0.979167 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2, score=0.980392 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2, score=0.941176 - 0.0s
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=0.1, features__univ_select__k=2, score=0.979167 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2, score=0.960784 - 0.0s
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=1, features__univ_select__k=2, score=0.979167 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2, score=0.980392 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2, score=0.921569 - 0.0s
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=2, svm__C=10, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1, score=0.980392 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1, score=0.941176 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=1, score=0.979167 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1, score=0.941176 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=1, score=0.979167 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1, score=0.921569 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=1, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2, score=0.980392 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2, score=0.941176 - 0.0s
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=0.1, features__univ_select__k=2, score=0.979167 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2, score=0.960784 - 0.0s
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=1, features__univ_select__k=2, score=0.979167 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2, score=1.000000 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2, score=0.921569 - 0.0s
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2
[CV] features__pca__n_components=3, svm__C=10, features__univ_select__k=2, score=1.000000 - 0.0s
[Parallel(n_jobs=1)]: Done 54 out of 54 | elapsed: 0.4s finished
Pipeline(memory=None,
steps=[('features', FeatureUnion(n_jobs=1,
transformer_list=[('pca', PCA(copy=True, iterated_power='auto', n_components=2, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)), ('univ_select', SelectKBest(k=2, score_func=<function f_classif at 0x7f4770b1bcf8>))],
transformer...,
max_iter=-1, probability=False, random_state=None, shrinking=True,
tol=0.001, verbose=False))])
在scikit-learn中,FeatureUnion是一个transformer,可以将多个transformer(例如Pipeline、ColumnTransformer等)组合起来并行执行,最终将它们的输出合并成单个特征空间。它的用法类似于Pipeline,但是Pipeline是串行执行,每个transformer只能使用前一个transformer的输出作为输入,而FeatureUnion是并行执行,每个transformer可以独立地使用原始输入,并将它们的输出合并在一起。
下面是一个简单的例子,展示了如何使用FeatureUnion将两个transformer组合在一起:
from sklearn.pipeline import FeatureUnion
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
# 创建两个transformer
scaler = StandardScaler()
pca = PCA(n_components=2)
# 创建FeatureUnion
feature_union = FeatureUnion([
('scaler', scaler),
('pca', pca)
])
# 创建Pipeline,将FeatureUnion作为第一个步骤
pipeline = Pipeline([
('feature_union', feature_union),
# ... 添加其他步骤 ...
])
# 使用Pipeline进行训练和预测
pipeline.fit(X_train, y_train)
y_pred = pipeline.predict(X_test)
在上面的例子中,FeatureUnion将StandardScaler和PCA组合在一起,并行地执行它们。它们的输出将被合并成单个特征空间,然后传递给Pipeline中的其他步骤。
FeatureUnion与Pipeline的主要区别在于它们执行transformer的方式。Pipeline是串行执行每个transformer,并将前一个transformer的输出作为下一个transformer的输入。而FeatureUnion是并行执行每个transformer,每个transformer可以独立地使用原始输入,并将它们的输出合并在一起。因此,FeatureUnion在需要对不同的特征使用不同的transformer时非常有用。Pipeline通常用于在特征预处理和模型拟合之间串联一系列transformer的情况。
FeatureUnion通过将多个transformer的输出按列连接(即水平堆叠)来合并它们的特征。具体地说,它将每个transformer的输出沿着水平方向堆叠起来,然后将它们按列连接起来,形成一个新的特征矩阵。特征矩阵的列数等于所有transformer的输出特征的总数。
例如,假设我们有两个transformer,它们的输出分别为(100, 5)和(100, 10),其中100是样本数,5和10是特征数。FeatureUnion将它们的输出沿着水平方向堆叠起来,得到一个(100, 15)的矩阵,其中15是5和10的总和。在这个新的特征矩阵中,第1到第5列对应于第一个transformer的输出,第6到第15列对应于第二个transformer的输出。
注意,由于FeatureUnion是并行执行transformer的,因此它要求每个transformer的输出特征数必须相同(即列数必须相同)。如果有不同数量的特征,可以使用ColumnTransformer将它们组合起来,然后将ColumnTransformer作为FeatureUnion的一个步骤来使用。此外,如果两个transformer的输出特征数相同,但特征的语义不同,那么在将它们合并成一个特征空间之前,需要对它们进行适当的预处理,以确保它们在统计上是可比的。
FeatureUnion并没有将特征进行简单的相加。相反,它将多个transformer的输出特征按列连接在一起,形成一个新的特征矩阵,其中每一列对应于一个transformer的输出特征。
举个例子,如果我们有两个transformer,一个是PCA,另一个是特征选择,它们的输出分别是(100, 5)和(100, 10),其中100是样本数,5和10是特征数。那么,FeatureUnion将它们的输出按列连接起来,形成一个新的(100, 15)的特征矩阵,其中15是5和10的总和。在这个新的特征矩阵中,前5列对应于PCA的输出特征,后10列对应于特征选择的输出特征。
因此,FeatureUnion并没有对特征进行简单的相加,而是将它们组合成一个新的特征空间。在这个新的特征空间中,每个特征都对应于一个transformer的输出特征。如果两个transformer的输出特征数不同,FeatureUnion将无法将它们连接在一起,因此需要确保每个transformer的输出特征数相同。

